Amazon SageMaker による結果の可視化
JupyterLabでの可視化
検索バーでsagemakerと入力し、候補のSageMakerをクリックします。
左側ナビゲーションペインからノートブックインスタンスをクリックします。
AgcResultNotebookが作成されていることを確認し、右側のJupyterLabを開くをクリックします。
ファイルエクスプローラー上部の上向き矢印のUploadボタンをクリックし、事前に共有したフォルダの中から、igv.ipynbをアップロードします。
ファイルエクスプローラーに表示されたら、ダブルクリックして表示をします。
2番目のセルのs3Baseのバケット名をAGC_BUCKETの値に書き換えます。
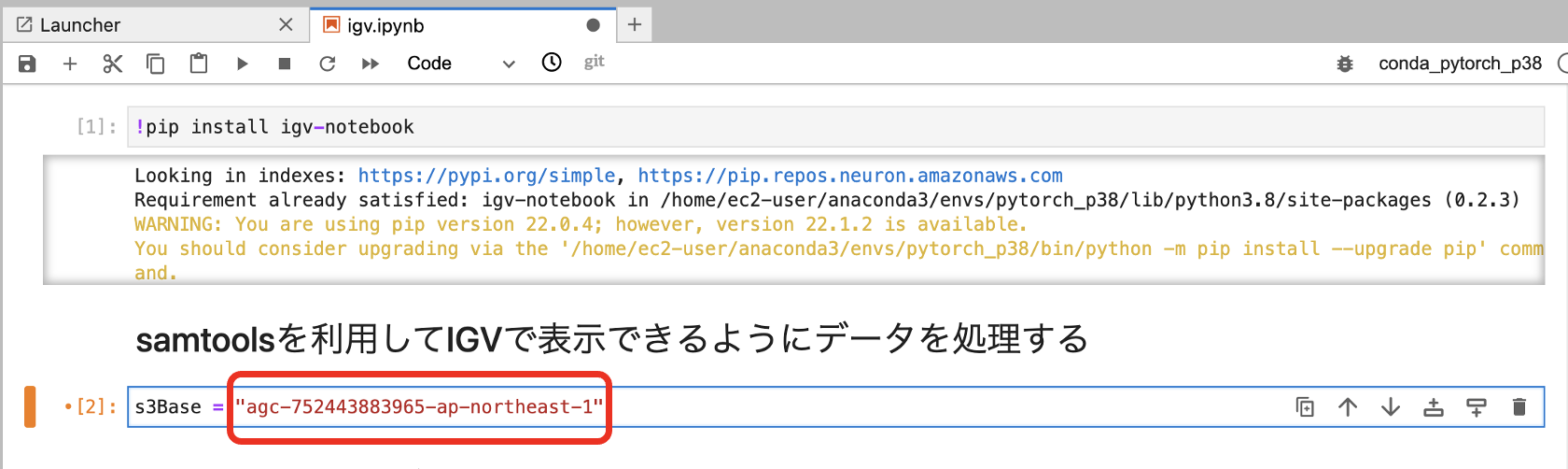
値がわからない場合は、Cloud9環境に戻り、以下を実行し、出力をコピーし、Jupyter Notebookへペーストします。
echo $AGC_BUCKET
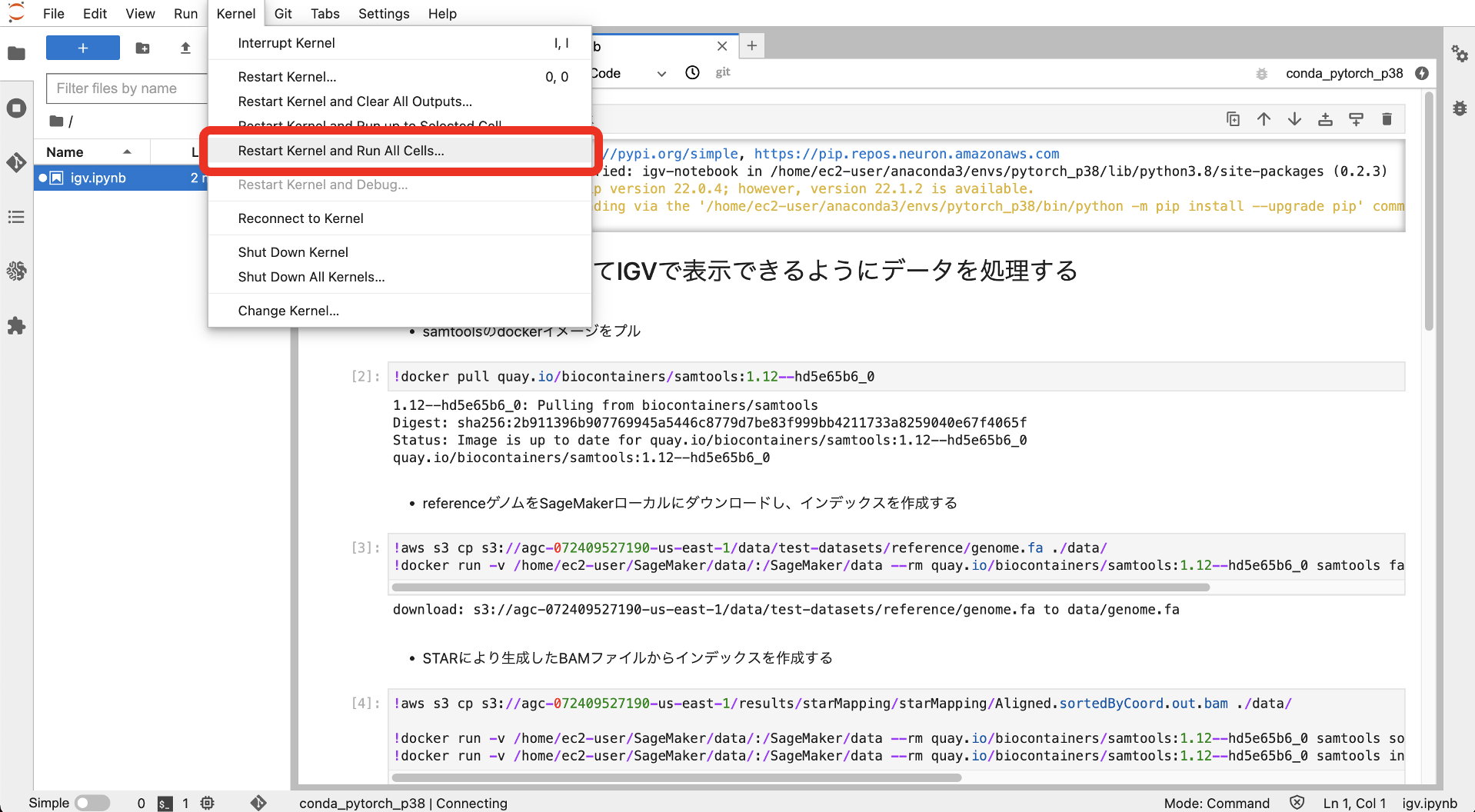
書き換えが完了したら、ノートブック上部のメニューバーからKernelを選択し、Restart Kernel and Run All Cellsをクリックします。
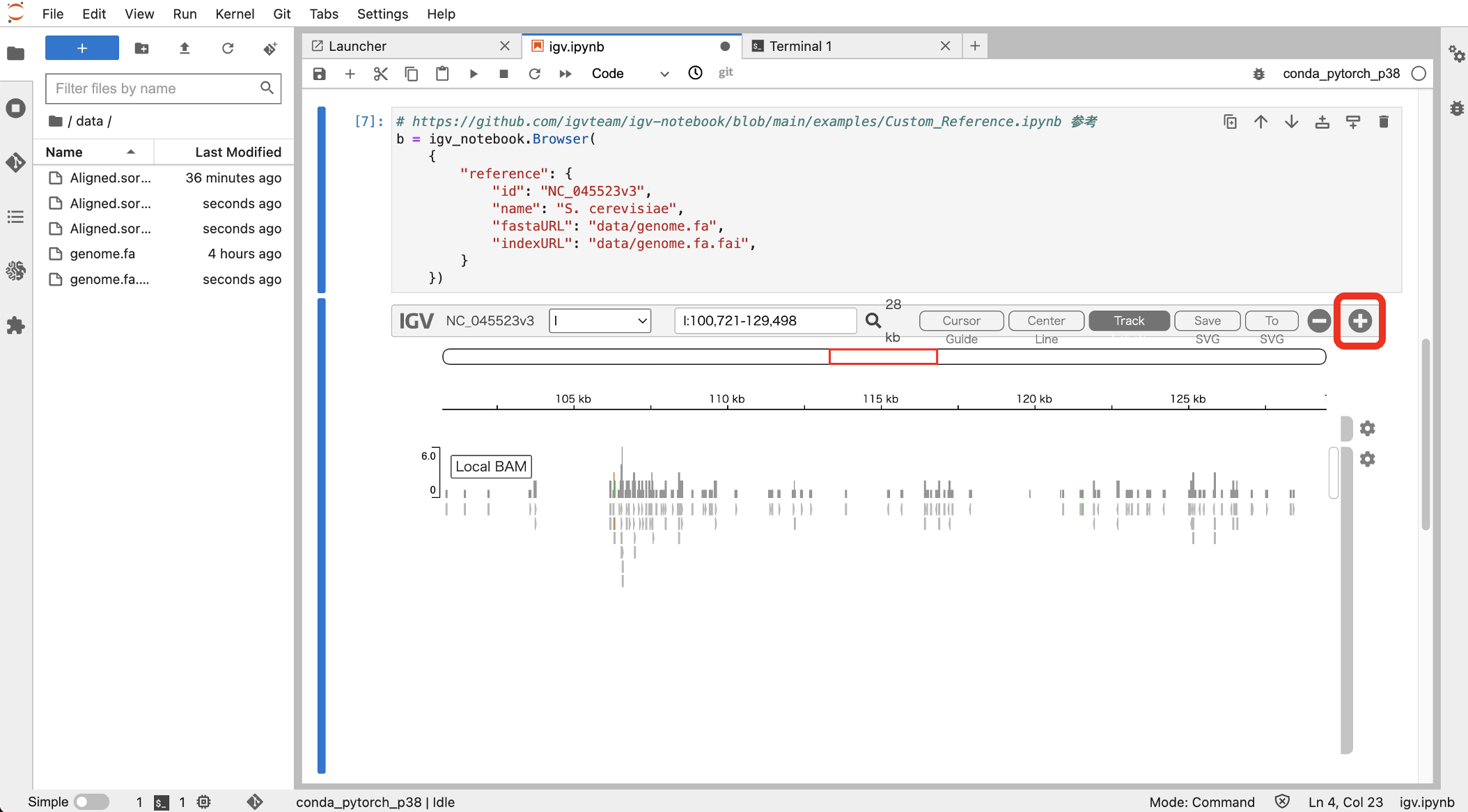
以下のようなIGVの描画結果がノートブック上で確認できたら成功です!Amazon Genomics CLIによる解析結果をクラウド上のJupyter Notebookで可視化できることが確認できました!
(注:表示が見えない場合には拡大してみてください)
本章では、簡易RNAseqワークフローを実行し、その手軽なスケーリングメリットを実感していただきました。 また、結果について、ローカル環境にデータをダウンロードすることなく、AWSクラウド上だけで解析が完結しうることを体感いただきました。
本ハンズオンの正規の手順はこれにて終了です。お疲れ様でした!