AWS Cloud9 のセットアップ
Amazon Genomics CLIおよびNextflowの開発環境として利用するAWS Cloud9および結果の可視化に利用するAmazon SageMaker NotebookをAWS Cloudformationによりセットアップします。
AWS Cloudformationによる環境作成
ここではAWS CloudFormationを利用し、ハンズオンでの開発環境を構築します。作成されるリソースは、AWS Cloud9、Amazon SageMaker Notebook、関連するIAMリソースです。
AWS CloudFormationコンソールにアクセスします。検索窓にcfnと入力し、ヒットしたサービスCloudFormationをクリックします。
スタックの作成をクリックします。
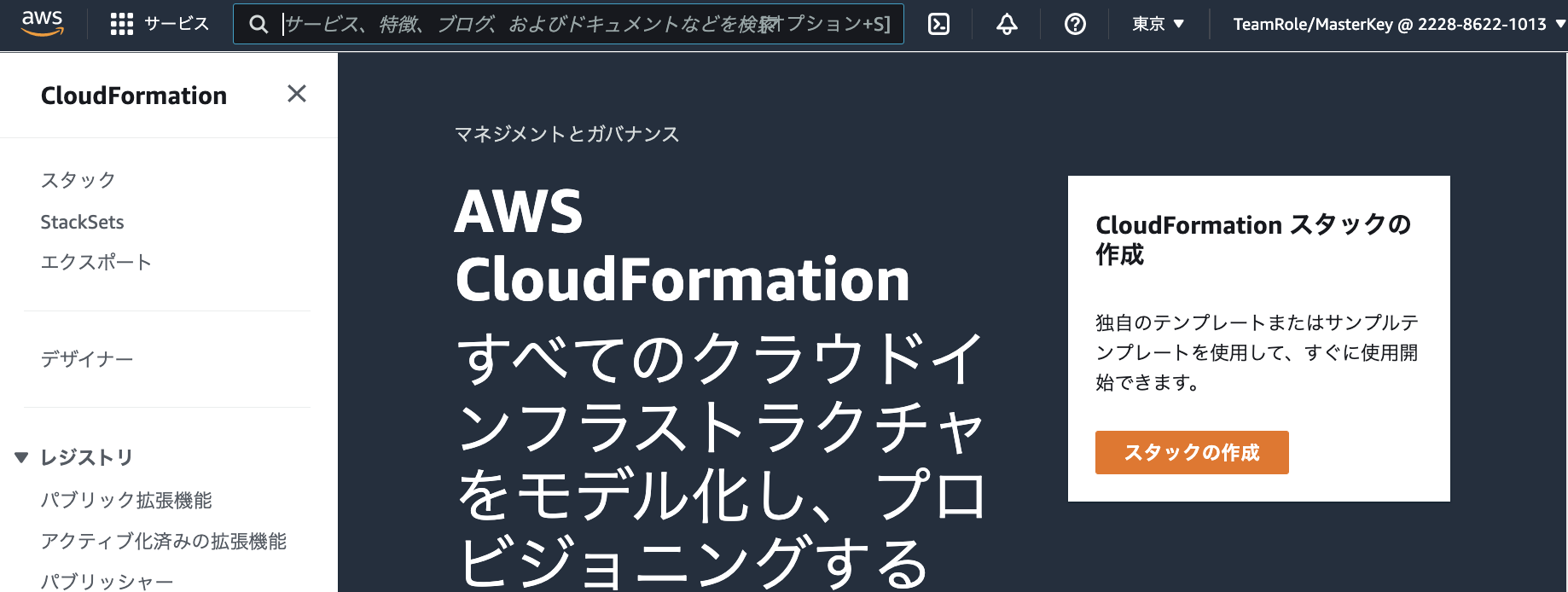
テンプレートの準備でテンプレートの準備完了が選択されていることを確認します。テンプレートの指定でテンプレートファイルのアップロードを選択し、ファイルを選択をクリックし、事前に共有したファイル群から、agc-handson-env.yamlを選択します。
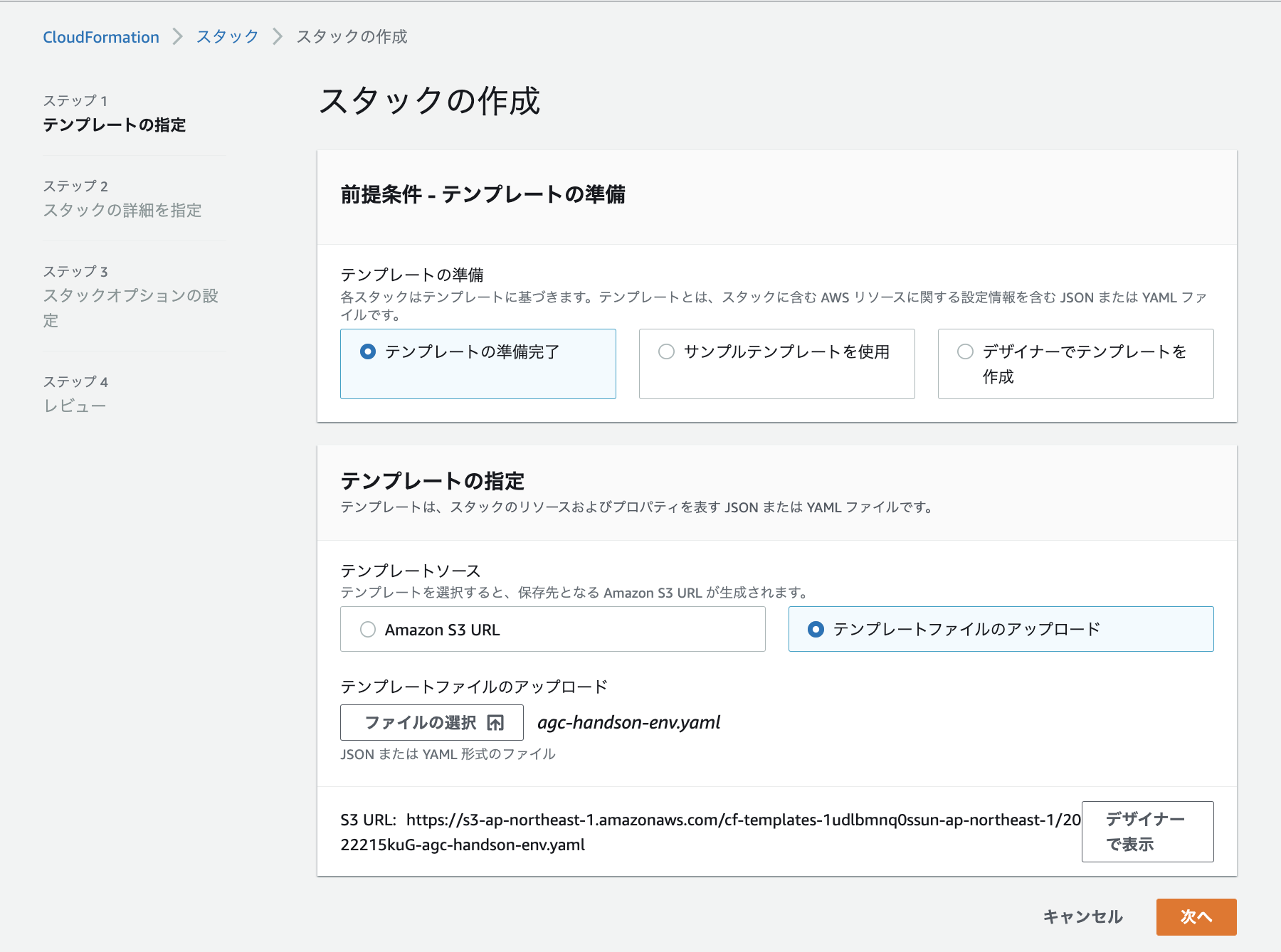
スタックの名前にはagc-handsonを入力します。残りの設定は変えずに次へをクリックします。
設定は変えずに、次へをクリックします。
以下のように表示されるチェックボックスにチェックを入れ、スタックを作成をクリックします。
以下のようにリソースの作成が開始します。
AWS Cloud9 コンソールへアクセス
マネジメントコンソールの検索窓に c9 と入力し、ヒットしたサービス Cloud9 をクリックします。
AWS Cloud9 環境の設定
インフラストラクチャーの設定
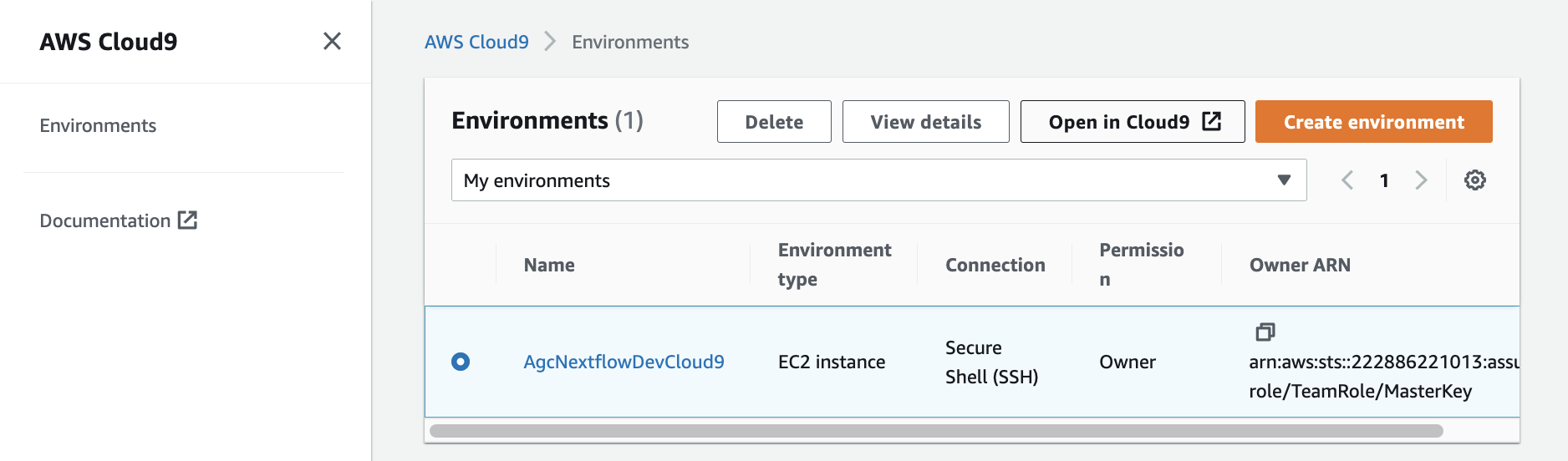
Environmentsより、AgcNextflowDevCloud9の左のチェックボックスにチェックを入れ、上部のOpen in Cloud9をクリックします。
以下のような画面が立ち上がります。
次に、Cloud9環境のIAM権限を設定します。
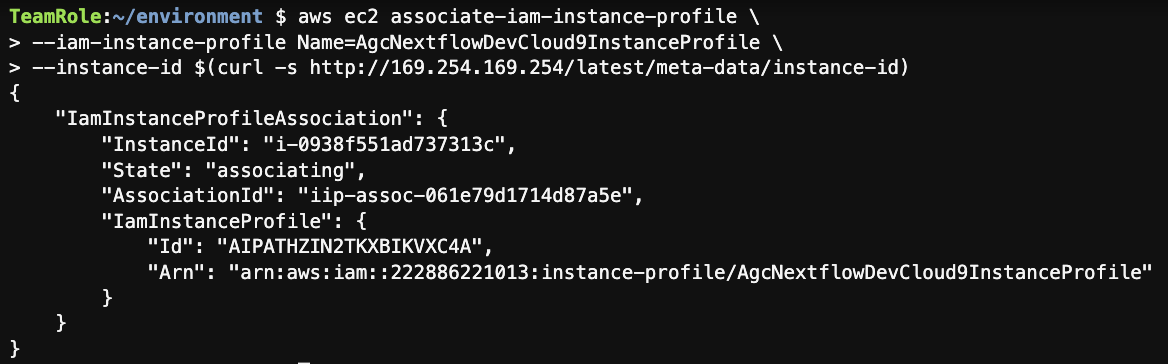
CloudFormationで作成したIAM InstanceProfileをCloud9に関連づけます。以下のコマンドを実行してください。
aws ec2 associate-iam-instance-profile \
--iam-instance-profile Name=AgcNextflowDevCloud9InstanceProfile \
--instance-id $(curl -s http://169.254.169.254/latest/meta-data/instance-id)
実行すると、以下のようなレスポンスが得られます。
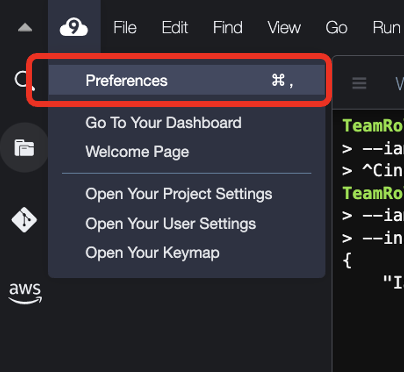
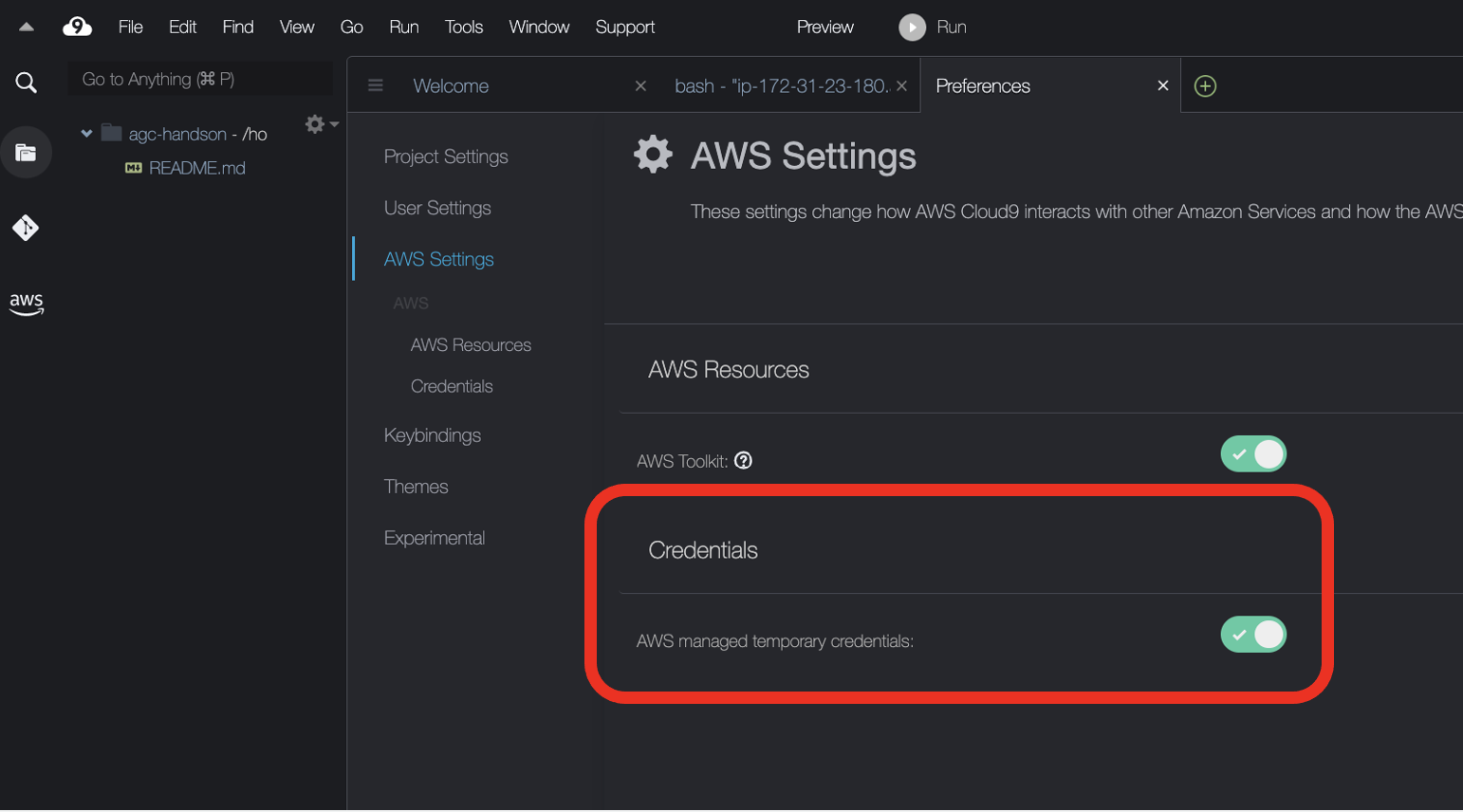
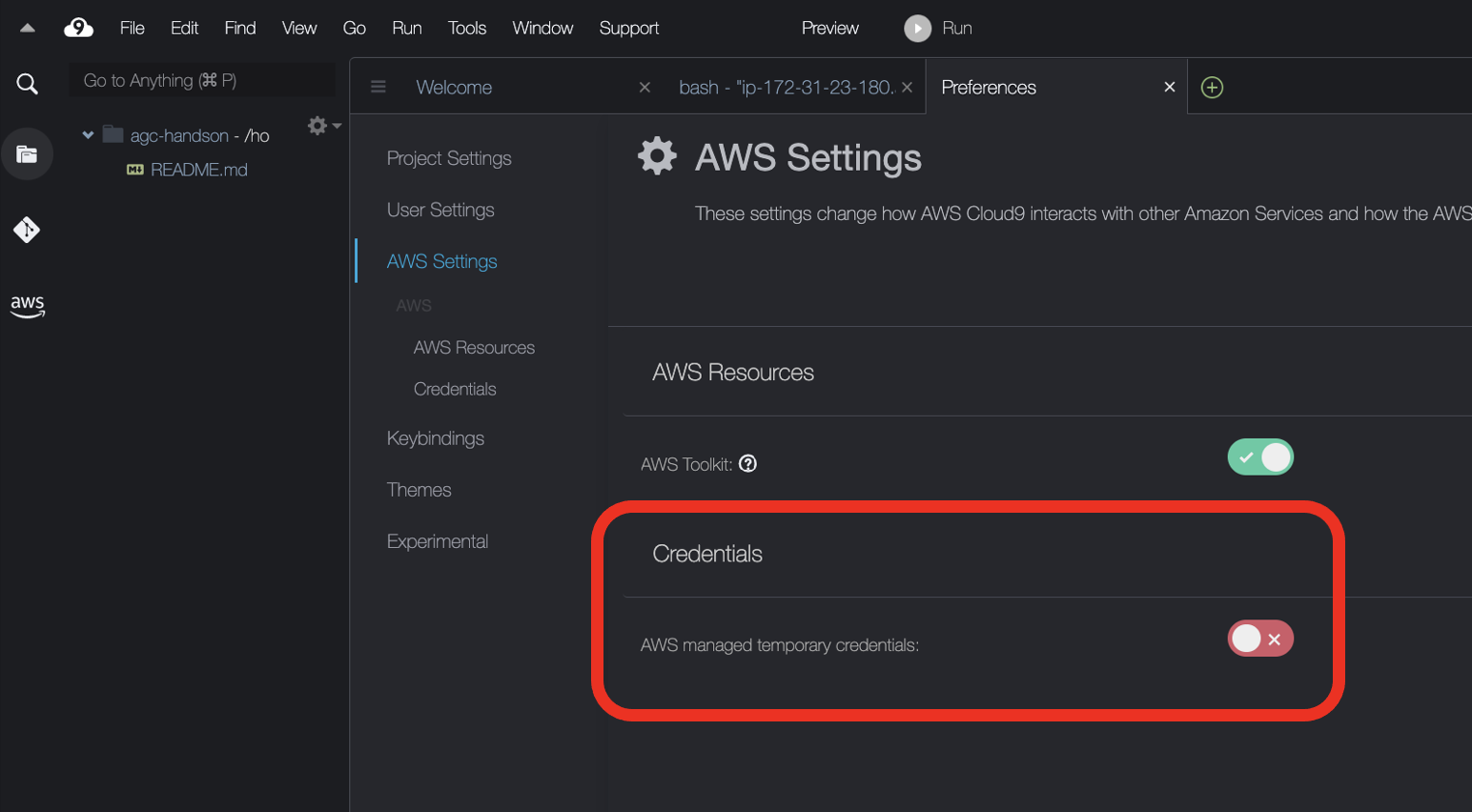
Cloud9環境の一時認証情報を無効化します。左上の雲のマークをクリックし、Preferencesを選択します。
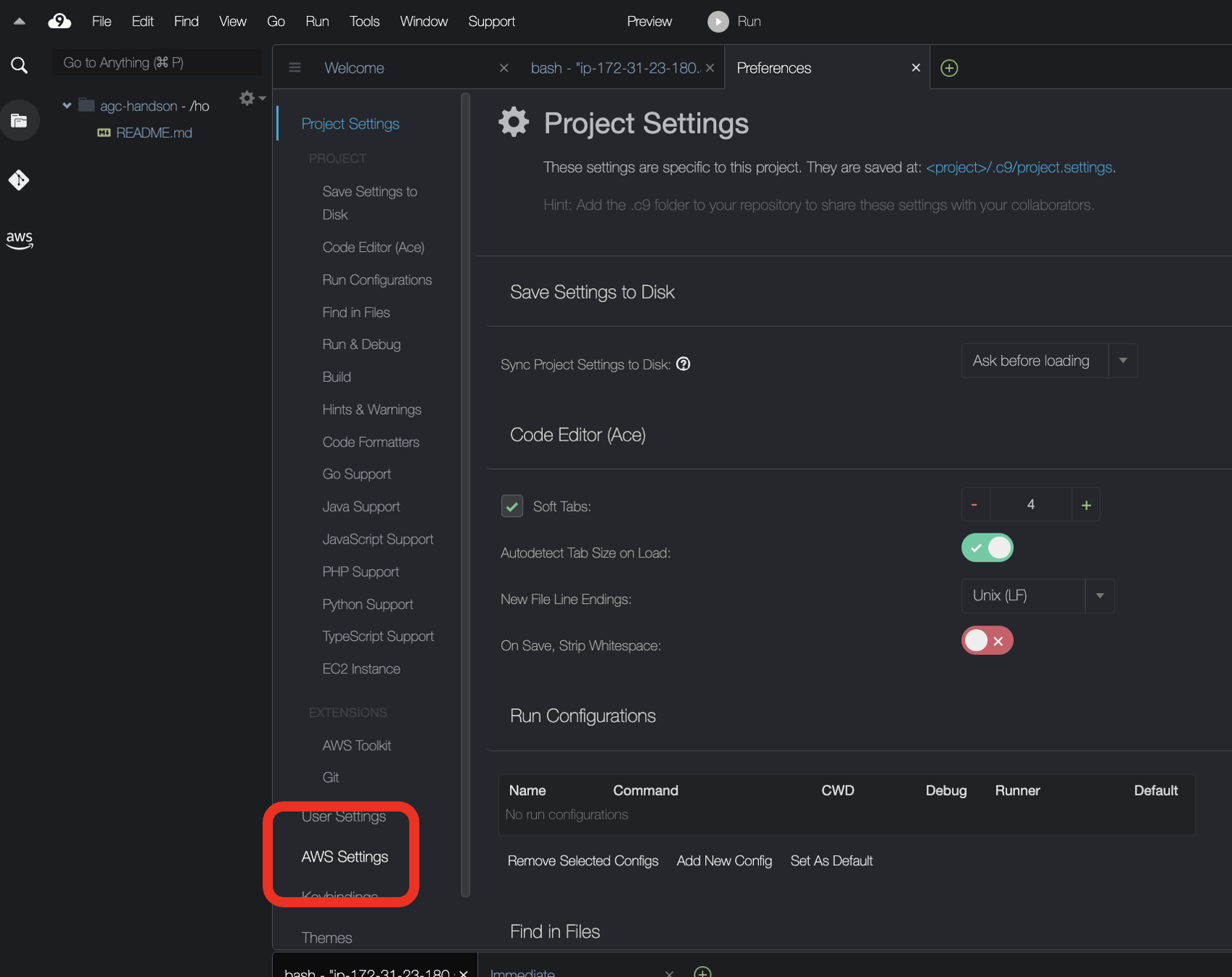
AWS Settingsをクリックし、Credentials以下のAWS managed temporary credentialsをオフにします。
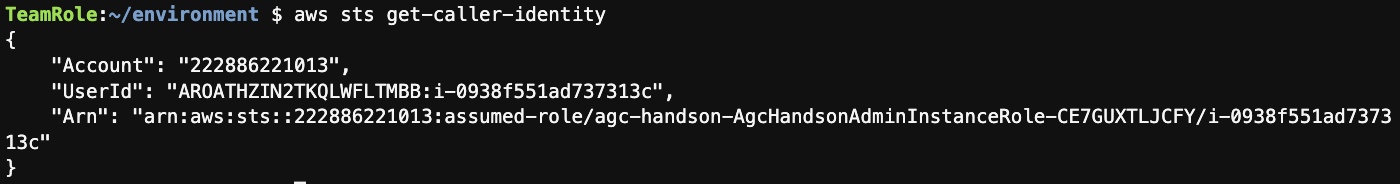
以下のコマンドを実行し、作成したIAM Roleがインスタンスに設定されていることを確認します。
aws sts get-caller-identity
以下のように、AgcHandsonAdminInstanceRoleを名前の一部に含むIAM Roleがアタッチされていれば成功です。
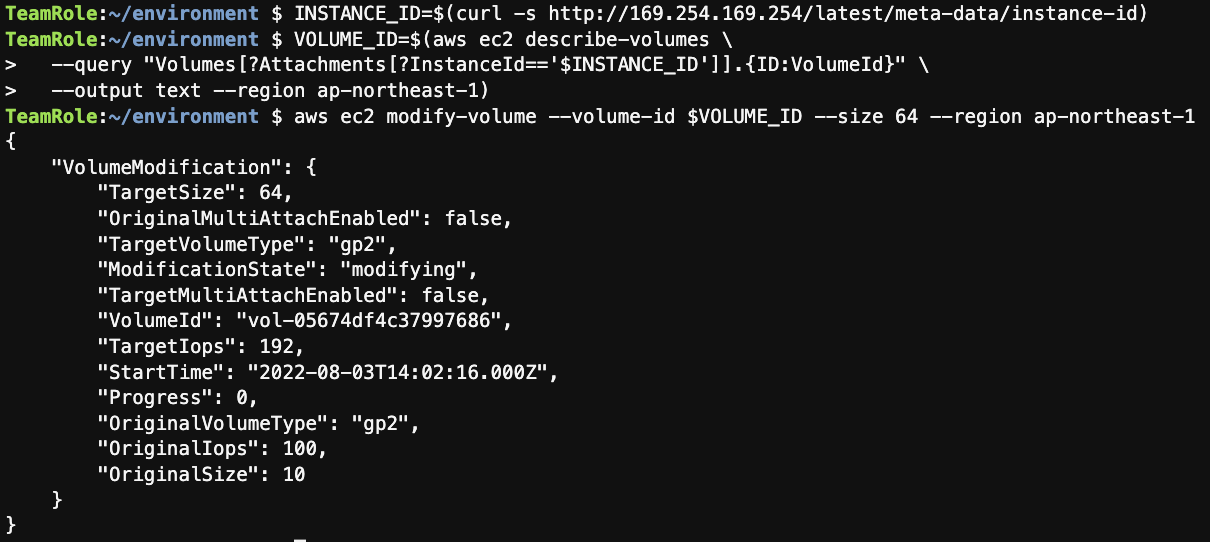
以後の解析のために、EC2インスタンスにアタッチされているEBSボリュームを拡張します。以下のコマンドを実行してください。
INSTANCE_ID=$(curl -s http://169.254.169.254/latest/meta-data/instance-id)
VOLUME_ID=$(aws ec2 describe-volumes \
--query "Volumes[?Attachments[?InstanceId=='$INSTANCE_ID']].{ID:VolumeId}" \
--output text --region ap-northeast-1)
aws ec2 modify-volume --volume-id $VOLUME_ID --size 64 --region ap-northeast-1
次のような実行結果が得られます。
ファイルシステムを拡張します。
sudo growpart /dev/nvme0n1 1
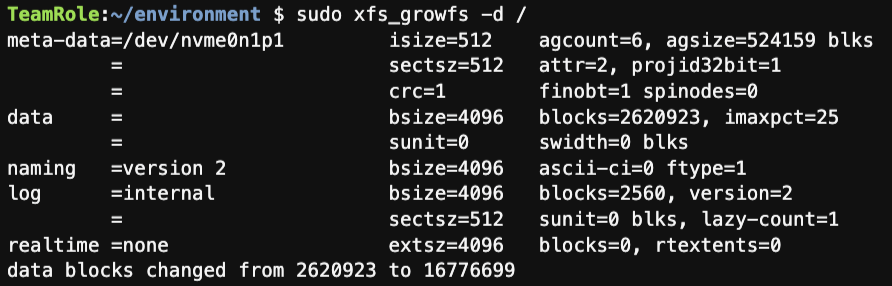
sudo xfs_growfs -d /
パーティションサイズが拡張できたことが確認できます。
 xfsファイルシステムのサイズが拡張できたことが確認できます。
xfsファイルシステムのサイズが拡張できたことが確認できます。
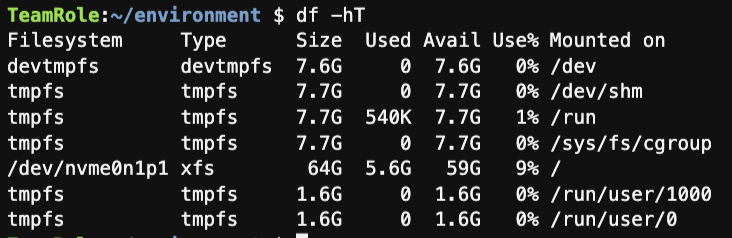
以下のコマンドでファイルシステムの拡張が行えたかどうかを確認してください。
df -hT
以下のようにファイルシステムのSizeが64Gに拡張できたことが確認できます。
パッケージのインストール
インフラストラクチャーの設定が終わったので、続いて、AWS CLIの環境変数設定を行います。
sudo yum install -y jq
export ACCOUNT_ID=$(aws sts get-caller-identity --output text --query Account)
export AWS_REGION=$(curl -s 169.254.169.254/latest/dynamic/instance-identity/document | jq -r '.region')
echo "export ACCOUNT_ID=${ACCOUNT_ID}" >> ~/.bash_profile
echo "export AWS_REGION=${AWS_REGION}" >> ~/.bash_profile
aws configure set default.region ${AWS_REGION}
aws configure get default.region
モジュール (Boto3) をインストールします。
pip3 install --user boto3
Nextflowをインストールします。
wget -qO- https://get.nextflow.io | bash
sudo mv nextflow /usr/local/bin
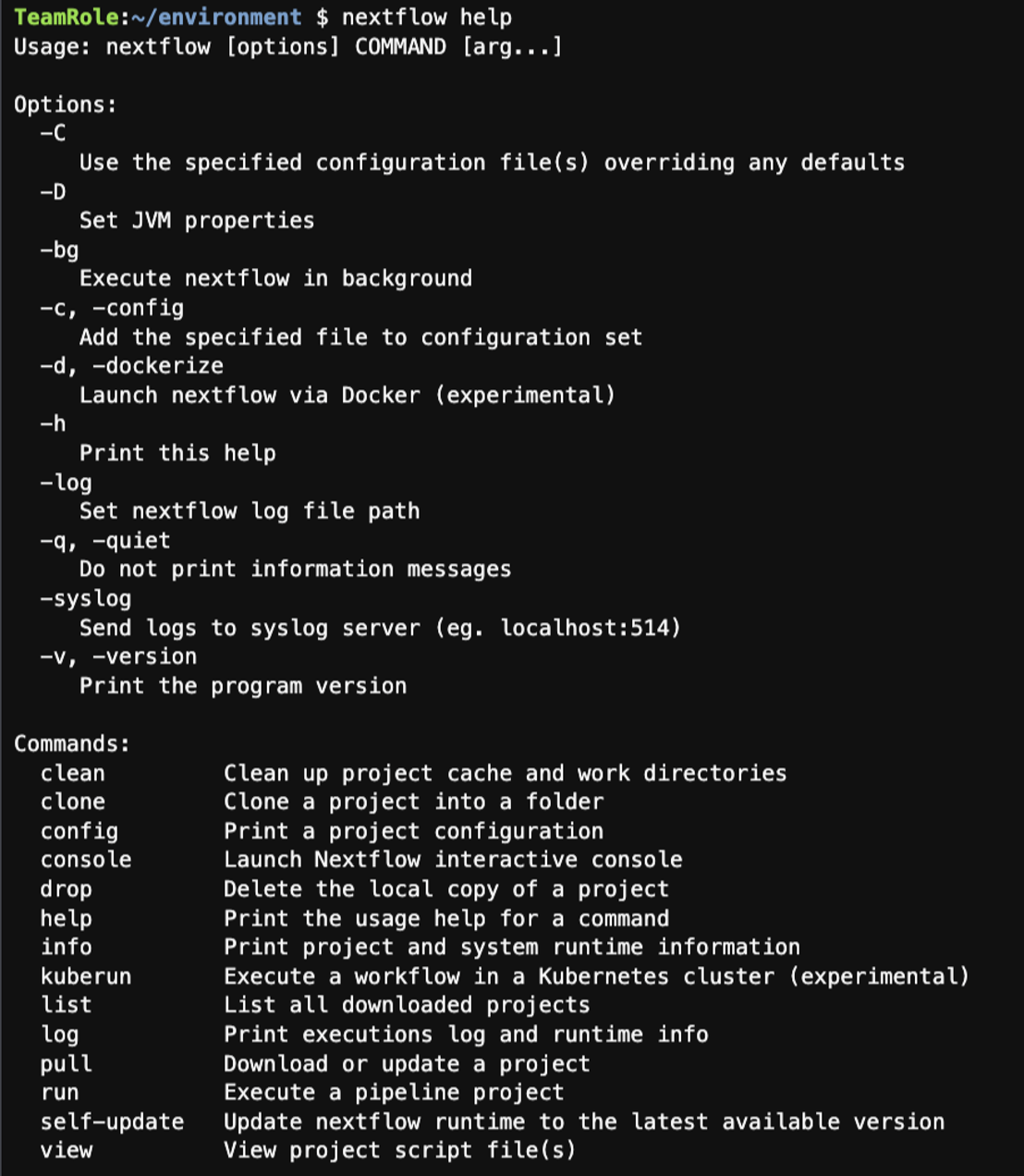
上記コマンドが完了したら、インストールが正常に行えたか、以下コマンドで確認します。
nextflow help
以下のようにヘルプ情報が得られればインストール成功です。
Amazon Genomics CLIをインストールします。
aws s3api get-object --bucket healthai-public-assets-us-east-1 --key amazon-genomics-cli/nightly-build/amazon-genomics-cli.zip amazon-genomics-cli.zip
unzip amazon-genomics-cli.zip
cd amazon-genomics-cli/
./install.sh
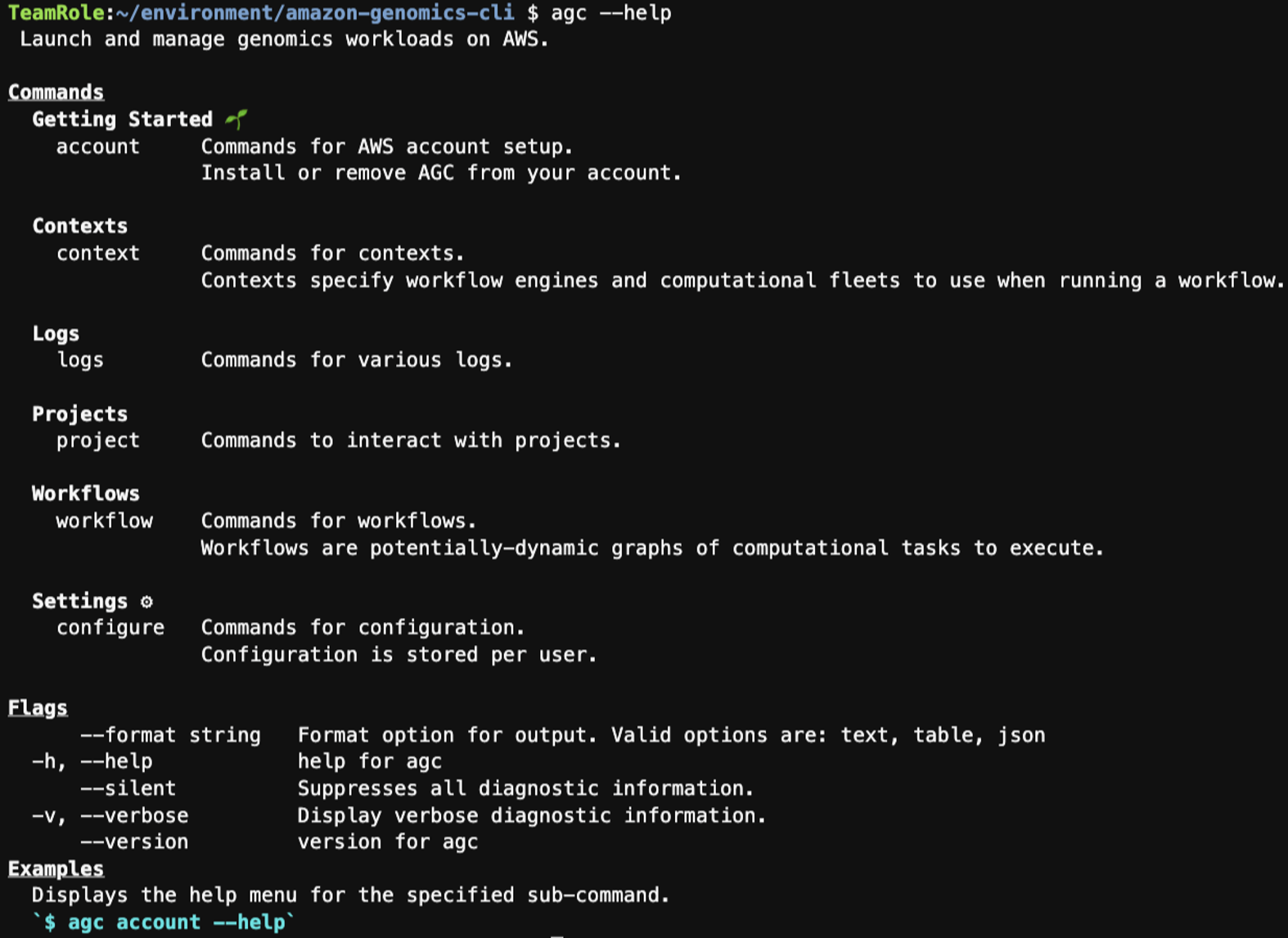
上記コマンドが完了したら、インストールが正常に行えたか、以下コマンドで確認します。
agc --help
以下のようにヘルプ情報が得られればインストール成功です。
利便性のため、コマンド補完を有効化します。
source <(agc completion bash)
Cloud9のターミナルでagcと入力したあと、タブを2回クリックするとサブコマンド候補が表示されることが確認できるはずです。
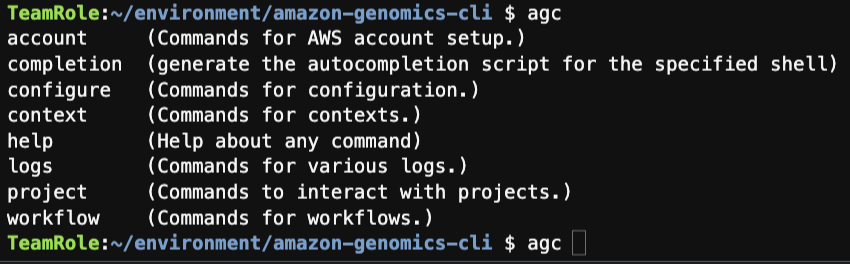
以上でAmazon Genomics CLIおよびNextflowをCloud9環境で実行できるようになりました。
次章で利用するデータのダウンロード
この章の最後に、次章で利用するRNAseqのデータはこの時点でバックグラウンドでのダウンロードを行っておきます。以下のコマンドを実行してください。
cd ~/environment/
mkdir data
cd data
git clone -b rnaseq https://github.com/nf-core/test-datasets.git &
jobsコマンドによりバックグラウンドでダウンロードのジョブが起動していることを確認します。

お疲れ様でした!これでAmazon Genomics CLIの開発環境が整いました。次章からいよいよAmazon Genomics CLIを利用してワークフローを実行していきます。